关于大样本量差异分析是否可以转为拟时序分析,以及两个分组的差异分析仅仅是上下调的问题,以下是经过伪原创处理后的文章:
许多朋友在后台表示对单细胞数据分析中的拟时序分析感到困惑。恰逢最近看到了一篇清晰展现拟时序分析重要性的文献,这里与大家分享。这篇文献完美地展示了为什么单纯的差异分析不足以揭示全部信息,而拟时序分析则是对差异分析的深入剖析。
这篇发表在NATURE COMMUNICATIONS | (2021) 的文章:《CD177 modulates the function and homeostasis of tumor-infiltrating regulatory T cells》,链接是:https://www.php.cn/link/1ee9bee2c7227c35ac1ca90f2e4fb172:
研究开始时使用了13,433个外周血单核细胞(PB)和12,239个肿瘤浸润细胞(TI),通过降维聚类分群后,根据FOXP3和CD25 (IL2RA)定位到了Treg亚群,分别是160个PB和574个TI Treg细胞。
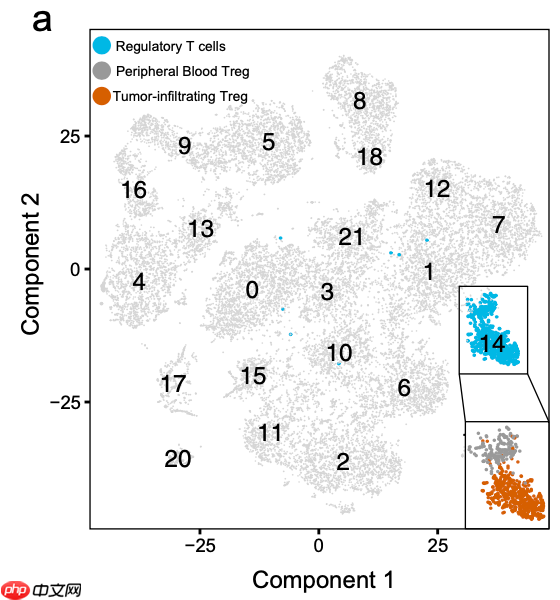 降维聚类分群
降维聚类分群
如果直接对这两个分组进行差异分析,可以识别出273个差异表达基因(DEGs)(Log fold-change > 1, adjusted p-value < 0.05)。
此外,作者在自己的ccRCC单细胞矩阵以及一个公共数据集HCC中也进行了类似的差异分析,并筛选出共有基因:
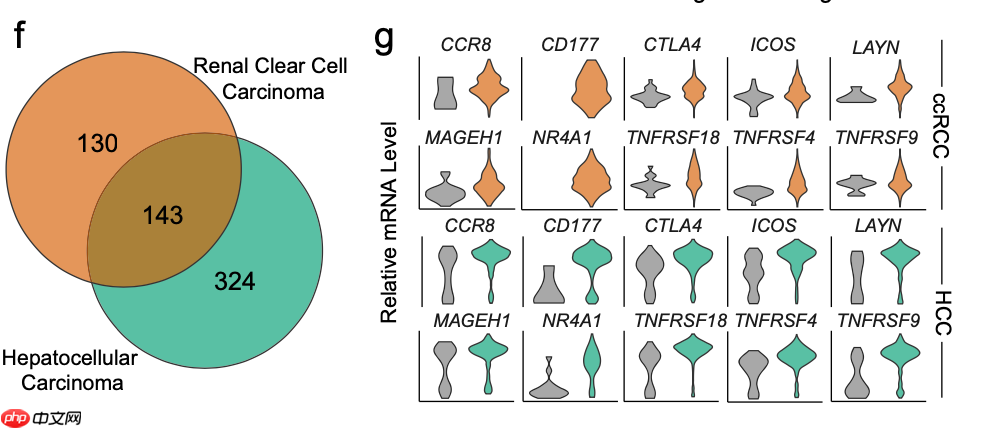 差异基因及其交集
差异基因及其交集
这样的差异分析虽然进行了交集筛选,但仍遗漏了许多细节,仅得到基因的上下调信息,而忽略了每个基因在两个单细胞亚群中具体的渐变趋势。
由于细胞数量并不多,运行Monocle 2算法,可以清楚地看到上下调的变化趋势,甚至发现隐藏的变化模式:
首先是拟时序分析的轨迹流形图,展示了ccRCC中Treg细胞的轨迹,使用Monocle 2算法。实线和虚线代表由表达谱定义的不同细胞轨迹/命运。
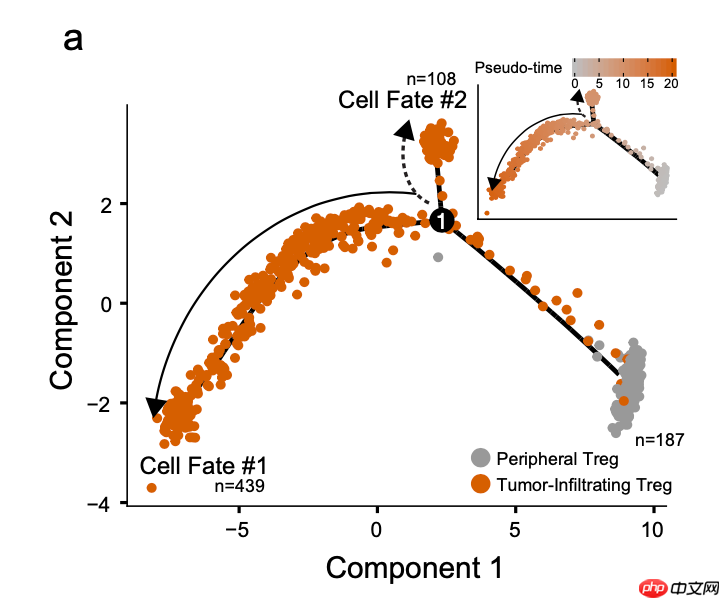 拟时序分析结果
拟时序分析结果
虽然拟时序分析只展示了上述的一个图,但具体的代码步骤还是有一定难度的。以下是可直接使用的代码示例,我们以SeuratData包中的pbmc3k数据集为例,主要是将Seurat包的对象转换为monocle中的单细胞对象:
代码语言:javascript
library(SeuratData) # 加载seurat数据集getOption('timeout')options(timeout=10000)#InstallData("pbmc3k")data("pbmc3k")sce <- as.SingleCellExperiment(pbmc3k[, 1:10, ])sc_cds <- importCDS(sce, import_all = TRUE)cds <- estimateSizeFactors(cds)cds <- estimateDispersions(cds)cds <- detectGenes(cds, min_expr = 0.1)cds = 10)))cds <- reduceDimension(cds, max_components = 2, method = 'DDRTree')cds <- orderCells(cds)save(cds, file = 'input_cds.Rdata')
确保前面的步骤无误后,接下来就可以运行拟时序分析的主程序:
代码语言:javascript
rm(list=ls())options(stringsAsFactors = F)library(monocle)library(Seurat)load(file = 'input_cds.Rdata')# 接下来很重要,到底是看哪个性状的轨迹colnames(pData(cds))table(pData(cds)$Cluster)table(pData(cds)$Cluster, pData(cds)$celltype)plot_cell_clusters(cds, 1, 2)## 我们这里并不能使用 monocle的分群# 还是依据前面的 seurat分群, 其实取决于自己真实的生物学意图pData(cds)$Cluster = pData(cds)$celltypetable(pData(cds)$Cluster)Sys.time()diff_test_res <- differentialGeneTest(cds, fullModelFormulaStr = "~Cluster")save(diff_test_res, cds, file = 'output_of_phe2_monocle.Rdata')
有了上面的output_of_phe2_monocle.Rdata文件后,就是拟时序分析的结果,接下来就可以进行各种可视化。首先看看每个细胞的时序信息:
 析稿Ai写作
析稿Ai写作
科研人的高效工具:AI论文自动生成,十分钟万字,无限大纲规划写作思路。
 97 查看详情
97 查看详情 
代码语言:javascript
## 后面是对前面的结果进行精雕细琢rm(list=ls())options(stringsAsFactors = F)library(Seurat)library(gplots)library(ggplot2)library(monocle)library(dplyr)load(file = 'output_of_phe2_monocle.Rdata')cds = my_cds_subsetcolnames(pData(cds))table(pData(cds)$State, pData(cds)$Cluster)library(ggsci)p1 = plot_cell_trajectory(cds, color_by = "Cluster") + scale_color_nejm()p1ggsave('trajectory_by_cluster.pdf')plot_cell_trajectory(cds, color_by = "celltype")p2 = plot_cell_trajectory(cds, color_by = "Pseudotime")p2ggsave('trajectory_by_Pseudotime.pdf')p3 = plot_cell_trajectory(cds, color_by = "State") + scale_color_npg()p3ggsave('trajectory_by_State.pdf')library(patchwork)p1 + p2 / p3
可视化结果如下:
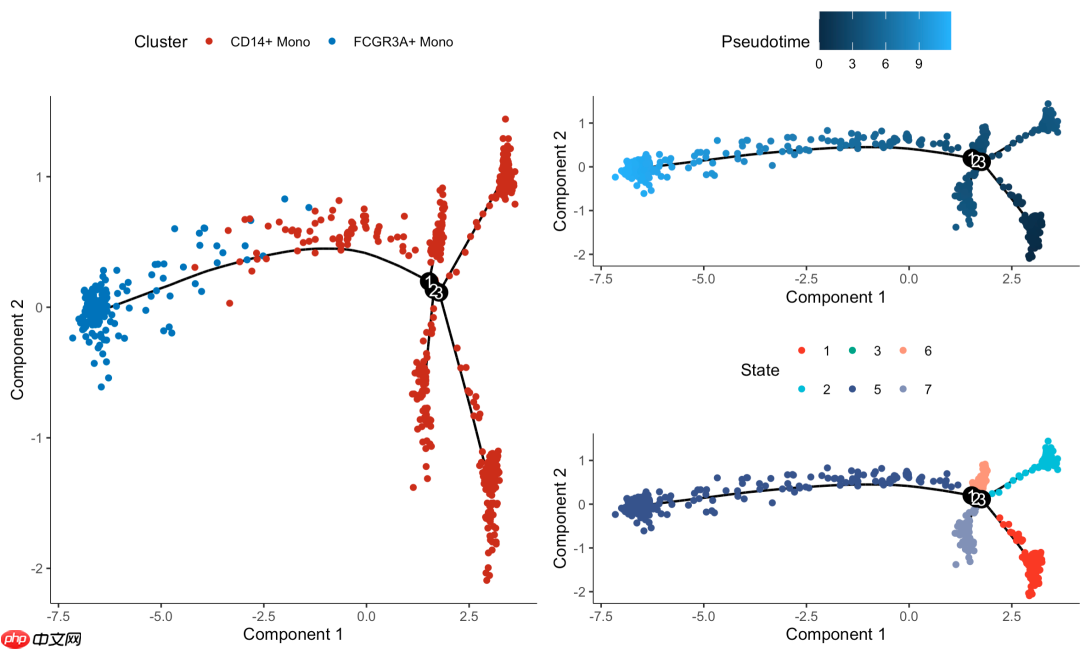 拟时序分析的多元化结果
拟时序分析的多元化结果
因为原文中恰好形成了两个细胞命运,所以可以使用BEAM函数,仍然是提取具体的基因进行热图可视化:
b 基于流形的免疫基因转录变化的拟时序投影。显著性基于相对于起始位置的差异测试,该起始位置也用于生成拟时序并进行了多重比较调整。
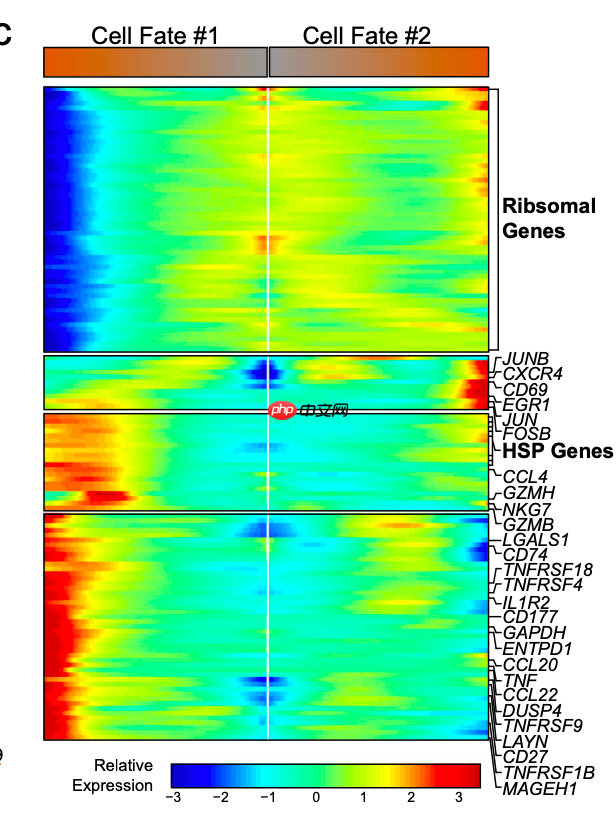 拟时序分析的差异基因热图
拟时序分析的差异基因热图
热图中的基因有多种展示方式:
c 显著基因(q < 0.05)的表达热图
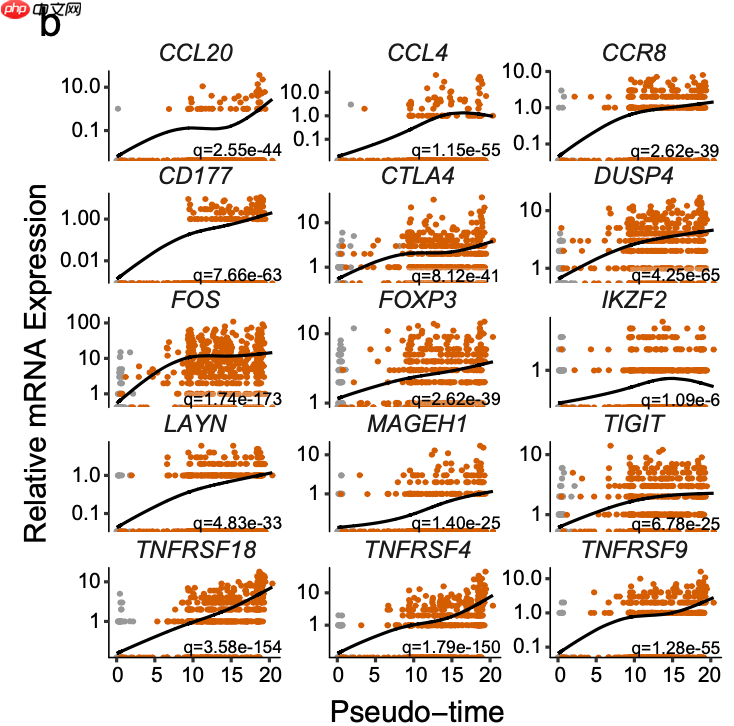 拟时序分析的差异基因趋势折线图
拟时序分析的差异基因趋势折线图
以及:
d 基于流形的免疫基因转录变化的细胞轨迹投影。显著性基于TI Treg细胞第一和第二细胞命运之间的差异测试。“x”表示流形两极的标尺化平均mRNA水平。
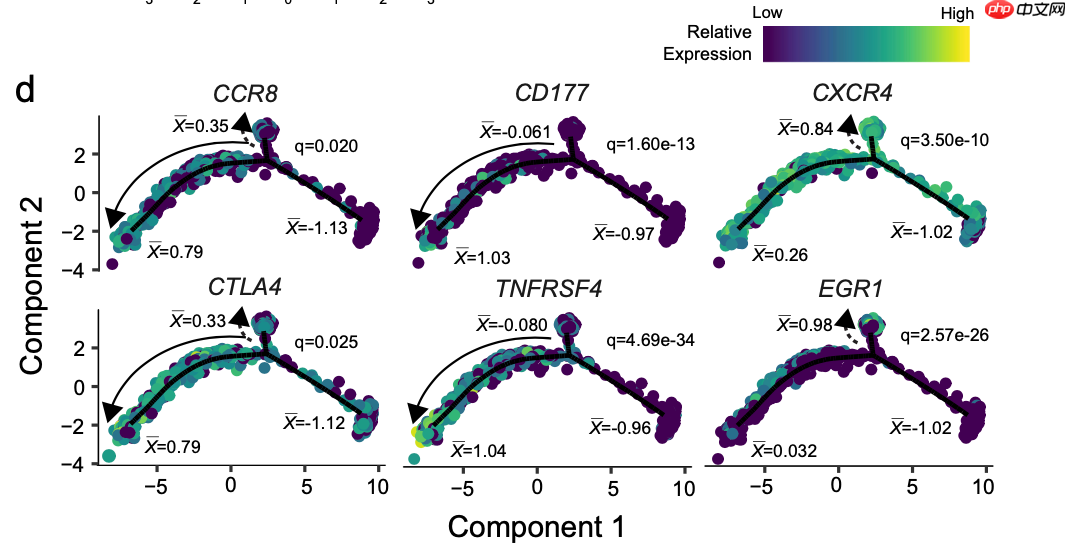 拟时序分析的差异基因表达量图
拟时序分析的差异基因表达量图
最后这个图,看起来技术含量十足!
通过以上内容可以看出,拟时序分析不仅可以揭示差异分析无法捕捉的动态变化,还能提供更细致的基因表达趋势和细胞命运信息。因此,大样本量差异分析转为拟时序分析可以提供更深入的洞察,而两个分组的差异分析不仅仅是简单地识别上下调基因,更重要的是理解这些变化的动态过程。
以上就是拟时序分析就是差异分析的细节剖析的详细内容,更多请关注创想鸟其它相关文章!
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。
如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 chuangxiangniao@163.com 举报,一经查实,本站将立刻删除。
发布者:程序猿,转转请注明出处:https://www.chuangxiangniao.com/p/472779.html









 微信扫一扫
微信扫一扫  支付宝扫一扫
支付宝扫一扫 


















